Restez toujours informé: suivez-nous sur Google Actualités (icone ☆)
Le cycle cellulaire est le processus par lequel le génome d'une cellule est dupliqué pour permettre la division et créer de nouvelles cellules. Il s'agit d'un processus fondamental, nécessitant un contrôle strict, les erreurs pouvant provoquer des pathologies tels que des cancers. Pour mieux en connaître le fonctionnement, une compréhension dynamique quantitative de la régulation des gènes tout au long du cycle fait encore défaut. L'outil DeepCycle développé ici apporte une méthode permettant de reconstruire des cartes d'expression génique du cycle cellulaire à haute résolution pour des modèles de cellules murines et humaines à différents stades du cycle.
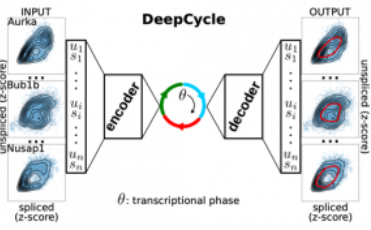
Actuellement, les outils utilisés pour observer la dynamique de l'expression des gènes lors du cycle cellulaire ne permettent pas de faire d'études in vivo, perturbent les cellules et sont peu généralisables. Pour apporter des données plus simples à interpréter, l'outil DeepCycle s'appuie sur un réseau de neurones profonds pour apprendre la trajectoire de l'expression des gènes lors du cycle cellulaire. Il analyse pour chaque gène la dynamique d'épissage des ARNm (non épissé/épissé) et en infère une "vitesse de l'ARN", valeur prédictive de l'état des cellules. De cette manière, DeepCycle permet de trier les cellules en fonction du stade du cycle cellulaire et de suivre leur progression, fournissant des données d'une très grande précision.
Pour démontrer la puissance de DeepCycle, les scientifiques ont généré des données unicellulaires de séquençage d'ARN en profondeur à partir de cellules souches embryonnaires de souris et de fibroblastes humains. Cela a permis de développer une dynamique d'expression génique à haute résolution, de découvrir des facteurs transcriptionnels coordonnant la régulation du cycle cellulaire et mettant en évidence le lien avec les régulateurs de pluripotence. Les scientifiques ont pu caractériser le point de branchement où les fibroblastes humains sortent de leur cycle cellulaire pendant la phase G1, et entrent en arrêt du cycle cellulaire (G0). Ils ont également identifié de nouveaux gènes marqueurs et facteurs de transcription à l'origine de ce processus, ouvrant la voie à la caractérisation systématique de la transition G1-G0 dans plusieurs modèles cellulaires.
Ainsi, DeepCycle constitue une avancée significative, en produisant des données plus robustes et plus généralisables, pour les champs de l'étude de la régulation du cycle cellulaire, de la dynamique de l'expression génique et de la recherche biomédicale en général.

DeepCycle est un auto-encodeur qui, à partir de lectures non épissées et épissées d'expériences de séquençage d'ARN unicellulaire, est capable de trier par ordinateur les cellules en fonction de leur progression tout au long du cycle cellulaire représenté par θ.
Pour en savoir plus:
Cell cycle gene regulation dynamics revealed by RNA velocity and deep-learning.
Riba A, Oravecz A, Durik M, Jiménez S, Alunni V, Cerciat M, Jung M, Keime C, Keyes WM, Molina N.
Nature Communications. 23 mai 2022 doi: 10.1038/s41467-022-30545-8.
Laboratoire:
Institut de génétique, biologie moléculaire et cellulaire-IGBMC (CNRS/Inserm/Université de Strasbourg) - 1, rue Laurent Fries. 76404 Ilkirch Cedex.
Contact:
Nacho Molina - Chercheur CNRS à l'Institut de génétique, biologie moléculaire et cellulaire - IGBMC (CNRS/Inserm/Université de Strasbourg) -
molinan at igbmc.fr
Populaires